CViS: Anwendungsbeispiele Forschung
Auf dieser Seite wollen wir Ihnen eine kleine Auswahl von Anwendungen in der Forschung präsentieren, die auf den Servern der virtual Desktop Infrastruktur laufen.
- Yasara - Dr. Johannes van den Boom, Molekularbiologie
- Gaussian und Gromacs - Dr. Maria J. Davila Garvin, Geologie
- Strand NGS - Dr. Manuela Wülling, Entwicklungsbiologie
- CellProfiler - Dr. Nina Schulze, ICCE, Imaging Centre Campus Essen
- Amber und Eigenentwicklung - Mike Nemec, Bioinformatics and Computational Biophysics
- Gromacs - Jean Noël Grad, Bioinformatics and Computational Biophysics
- Gnu R - Dr. Anja Lange – Bioinformatics and Computational Biophysics
- Gnu R - Dr. Manfred Jensen - Biodiversität
Dr. Johannes van den Boom, Molekularbiologie I
„Die VMs kann man rund um die Uhr rechnen lassen. Und egal ob man im Labor, von zu Hause oder im Büro arbeitet, man loggt sich einfach von dort ein und muss nichts mehr hin- und herkopieren.“
Anwendung: Yasara
Darstellung, Simulation und Modellierung molekularer Strukturen und Funktionen
Windows-VM: 10 Cores, 16 GB RAM, 1 GB Grafikkarte (vGPU mit NVIDIA M60)
Dr. Maria J. Davila Garvin, Geologie
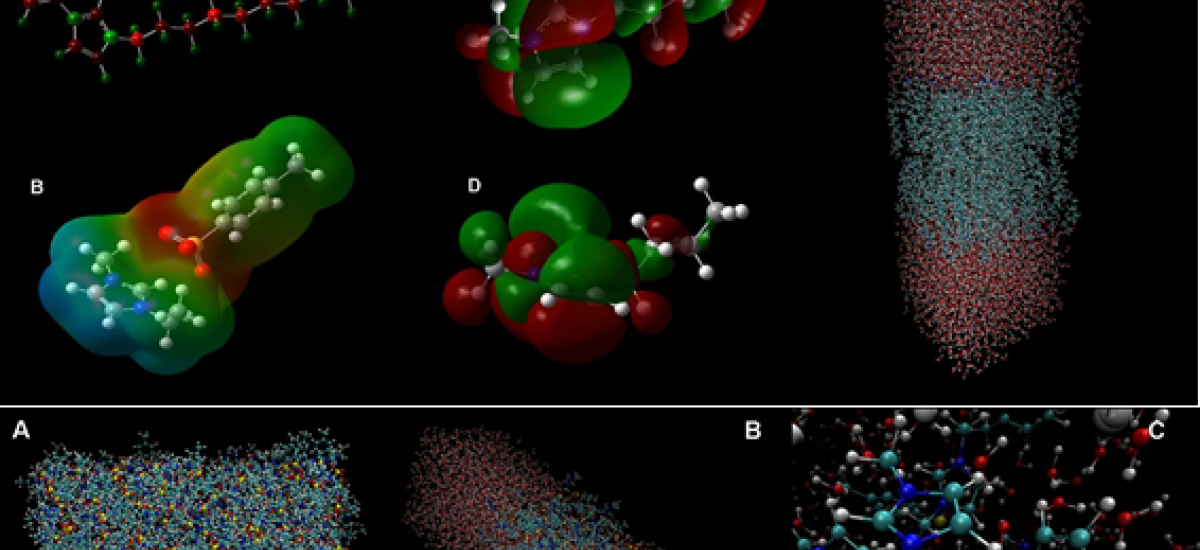
„My current research focuses on the synthesis of ionic liquids and the formation of self-assembled structures such as bilayer membranes. Through the use of computer simulations based on quantum mechanics and molecular dynamics in a virtual machine of the CViS, it is possible to analyse the molecular structure (ion-pair quantum calculations) and, to predict thermophysical properties and phase equilibrium conditions of systems containing ionic liquids (Fig. 1A-D and Fig. 2A-C). Another aspect of my research is to calculate the diffusion and permeability of solutes through bilayer systems as a function of pressure, temperature and composition (Fig. 1E), especially those formed in a two-phase system (supercritical CO2-liquid water).“
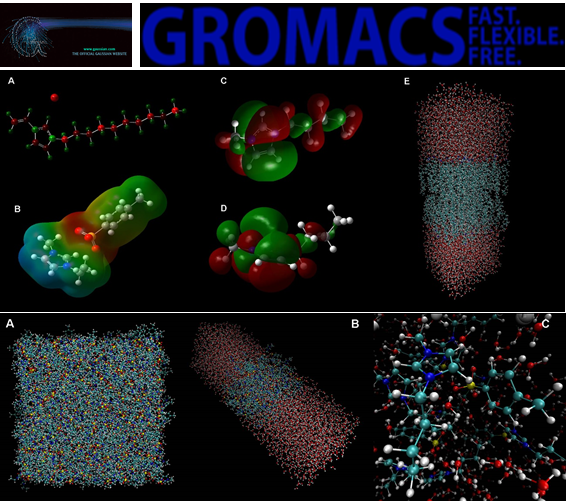
Anwendungen: Gaussian und Gromacs
Analyse molekularer Strukturen und Berechnungen von thermo-physikalischen Eigenschaften von Molekülen
Linux-VM (Ubuntu 64Bit/Eng): 10 Cores, 64 GB RAM
Dr. Manuela Wülling, Entwicklungsbiologie
„Wir untersuchen die Bindung verschiedener Proteine an DNA. Die NGS Software ermöglicht den Vergleich von Sequenzierungsdaten, um Bindungsprofile in unterschiedlichen Zelltypen zu vergleichen. Außerdem bietet sie Zugriff auf verschieden biologische Datenbanken, um Informationen über die identifizierten Regionen abzurufen.
Durch die Nutzung einer VM können wir die vorhandenen Einzelplatzlizenz sehr flexibel in der gesamten Arbeitsgruppe nutzen.“

Anwendungen: Strand NGS
Vergleichende Analyse von DNA-Sequenzierungen
Windows7-VM (64Bit): 8 Cores, 32 GB RAM
Dr. Nina Schulze, ICCE – Imaging Centre Campus Essen
„Die quantitative Bildanalyse ist einer der Schwerpunkte der Imaging Core Facility ICCE. Insbesondere für die automatisierte Analyse komplexer und großer Datensätze, wie sie beispielsweise bei high content screens entstehen, verwenden wir die Open-Source-Software CellProfiler (http://cellprofiler.org/). Die optimierte Konfiguration der VM ermöglicht es uns, solche Datensätze schnell, parallel und völlig unabhängig von der eigenen Hardware zu bearbeiten und so den Durchsatz signifikant zu erhöhen.“
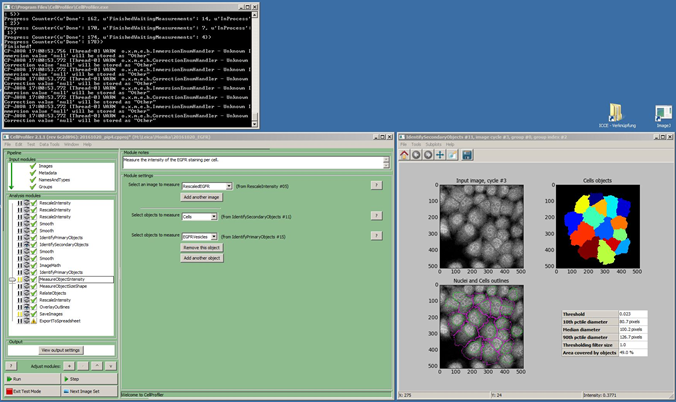
Anwendungen: CellProfiler
Quantitative Bildanalyse
2 x Windows7-VM (64Bit): 8 Cores, 16 GB RAM
Mike Nemec, Bioinformatics and Computational Biophysics
„Mittels MolekülDynamik Simulationen ist es möglich, Biomoleküle/Proteine auf atomarer Ebene zu untersuchen. Hierzu werden hunderttausende von 3D Strukturen der Moleküle durch Simulationen erstellt. Dies ist ein sehr rechenaufwendiger Prozess, bei dem alle Wechselwirkungen zwischen allen Atomen für jeden Simulationsschritt berechnet werden. Durch dynamische Allokation von mehreren CPUs und GPUs auf den virtuellen Maschinen kann die Berechnung parallelisiert werden. Dies wird von der Software zur Simulation Amber14 (ambermd.org) unterstützt.
Die riesige Anzahl an simulierten Strukturen führen zu Distanzmatrizen mit mehr als 100000x100000 Einträgen und können dank Zusammenschluss von viel Arbeitsspeicher parallel und effizient berechnet werden. Dadurch ist es möglich alle Molekülzustände miteinander zu vergleichen, und Rückschlüsse auf die Funktionsweise des Systems zu ziehen. Eigene Software (https://github.com/MikeN12/PySamplingQuality) wurde zur Analyse entwickelt, die vom dynamischen Zusammenschluss mehrerer CPUs in der virtuellen Maschine stark profitieren, um die große Anzahl von Simulationen und Strukturen parallel zu analysieren.“
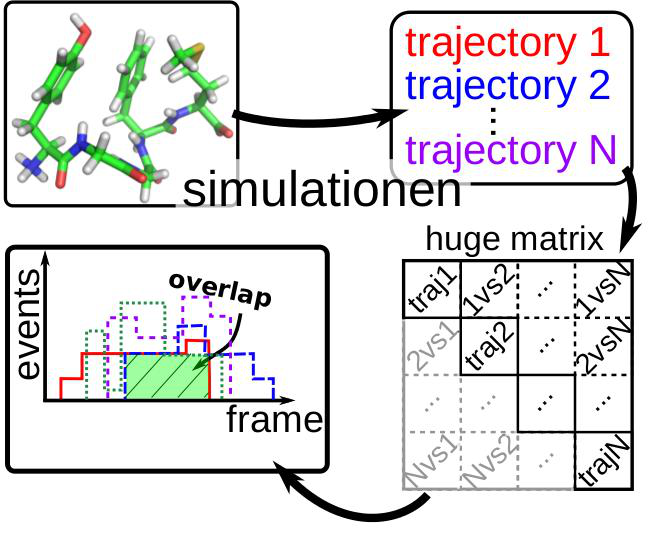
Anwendungen: Amber und selbstentwickelte Software
Klassifizierung der Qualität von MolekülDynamik - Simulationen von flexiblen Biomolekülen
Linux-VM (Ubuntu 64Bit/Eng): 16 Cores / 128 GB RAM
Jean Noël Grad, Bioinformatics and Computational Biophysics
„We focus our research on proteins involved in cell replication to better understand their function and explore new therapeutic strategies against cancer. GPU acceleration with 3 graphic cards on the same VM enables microsecond molecular dynamics simulations of complex proteins assemblies in a matter of weeks; the same calculations on our own GPU-enabled computers typically take months. These simulations allow us to predict the effect of synthetic drugs on proteins used by cancer cells to spread."
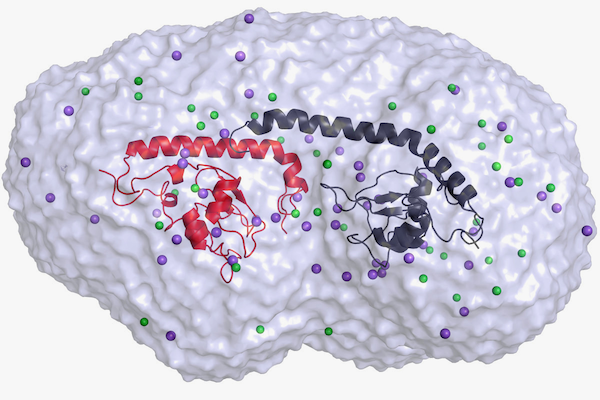
Figure: Molecular Dynamics von Survivin Monomere in Water/NaCl-suspension
Anwendungen: Gromacs
Moleculedynamic simulations of complex proteins assemblies
Linux-VM (Ubuntu 64Bit/Eng): 20 Cores / 128 GB RAM mit „direct pass through“ auf 3 K2 GPUs (über CUDA)
Dr. Anja Lange – Bioinformatics and Computational Biophysics
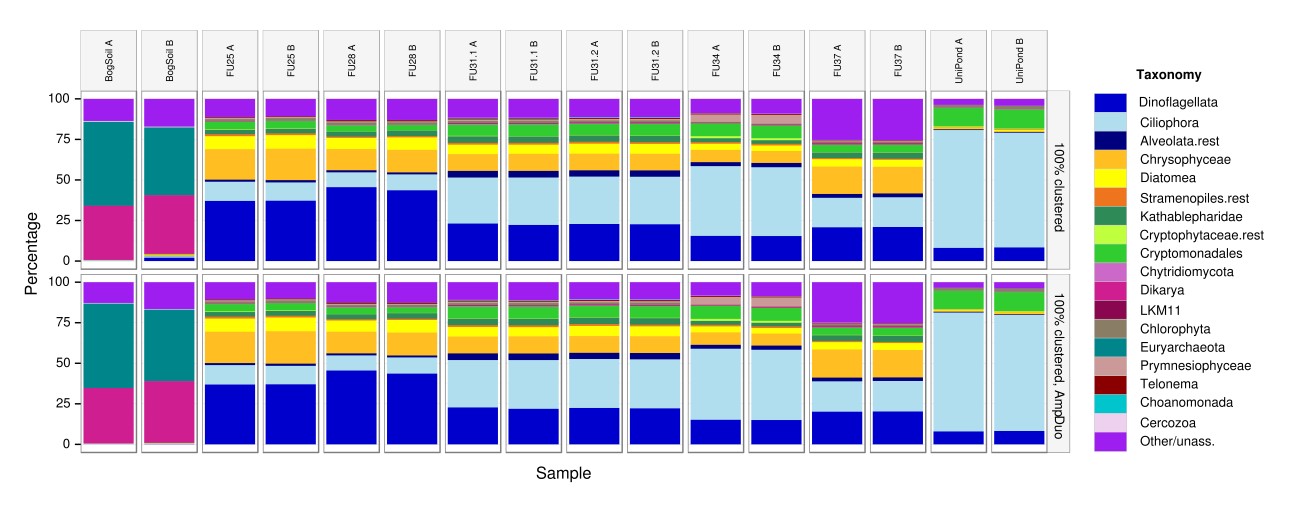
1) „Wir haben in Zusammenarbeit mit der AG Biodiversität eine Pipeline (primär basierend auf R und einigen anderen Programmen) zur Auswertung von Amplicon Daten entwickelt. Der Fokus in diesem Projekt liegt auf der Analyse der Diversität in unterschiedlichen Gewässern. Dazu wird aus Gewässerproben DNA isoliert, und spezifische Abschnitte des rRNA Gens mittels High Throughput-Sequenzierung (HT-Seq) sequenziert. Über die gefundenen rRNA Sequenzen kann auf die in den Gewässern vorhandenen Spezies geschlossen werden.
Mittel HT-Seq werden Millionen von Reads generiert, die Auswertung kann durch Parallelisierung deutlich beschleunigt werden, vor allem auch beim lokalen Blasten (BLAST+) gegen die Nukleotiddatenbank zur Zuordnung der Sequenzen zu Species. Die folgende Abbildung zeigt das Ergebnis einer solchen Analyse. Dargestellt ist die taxonomische Zuzsammesetzung verschiedener Proben von Seen und einer Bodenprobe (BogSoil), wobei alle Proben in Duplikaten analysiert wurden.“
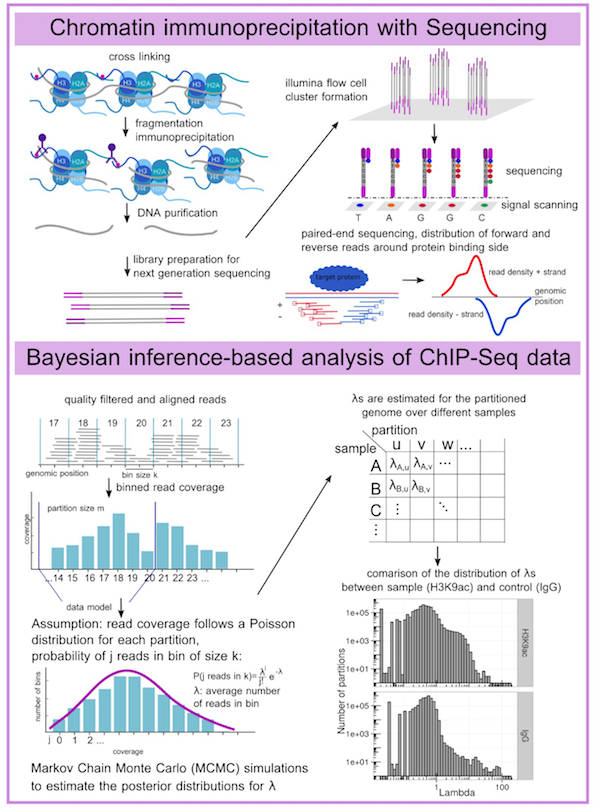
2) „Mit der AG Entwicklungsbiologie arbeiten wir an einem Projekt zur Charakterisierung von Histonmodifikationen unterschiedlicher Zelltypen im Maussystem. Das Prinzip von ChIP-Seq zur Identifizierung von Protein-DNA-Interaktionen ist in der Abbildung dargestellt. In vivo werden die Protein-DNA Komplexe reversible cross-linked, die DNA wird fragmentiert und die Protein-DNA Komplexe werden über Immunopräzipitation angereichert. Die DNA-Fragmente werden aufgereinigt und mittels HT-Seq sequenziert.
Die sequenzierten Reads müssen in einem ersten Schritt zum Mausgenom aligniert werden, dies ist paralleliesierbar und profitiert von der goßen Anzahl an CPUs in den VMs. Ein Ressourcen-kritischerer Schritt ist allerdings die spätere Auswertung der Signale. Zur Berechnung der Verteilungs-Parameter der Chip-Seq Reads benutzen wir Bayesian-inference mit dem Markov-Chain-Monte-Carlo-Verfahren (MCMC). Diese Methode benötigt sehr viel Arbeitsspeicher (120Gb RAM bei 16 Kerne, Laufzeit 4 Tage), was keiner unserer anderen Rechner zur Verfügung stellt.“
Anwendungen: R
Diversitätsanalyse von Gewässern durch DNA-Probenahmen
Analyse von Protein-DNA-Interaktion über ChIP-Seq
Linux-VM (Ubuntu 64Bit/Eng): 16 Cores / 128 GB RAM
Dr. Manfred Jensen - Biodiversität
„Ich schätze meine VM Maschinen sehr, nicht nur wg. ihrer Größe und phänomenalen Zuverlässigkeit (eigene Wartung entfällt), auch bei längeren Jobs (>14 Tage, das ist nicht selbstverständlich!), sondern auch wegen ihrer Flexibilität. Bei mir sind viele R-Scripte sowohl unter Linux als auch unter Windows lauffähig, wobei beide VM-Maschinen auf dieselben Verzeichnisse und dieselben Daten-Dateien zurückgreifen können. Für mich ganz zentral: die Script Sprache R und RStudio, insbesondere für multivariate statistische Auswertungen aller Art, z.B. NMDS, distance-based-RDA, Clusteranalyse, Diskriminanzanalyse, Netzwerkanalyse. Dies ist auch mit großen Tabellen möglich, bei denen lokale Maschinen aussteigen. Im Beispiel auch mal eine kleinere geographische Auswertung mit farbiger Cluster-Zuordnung.
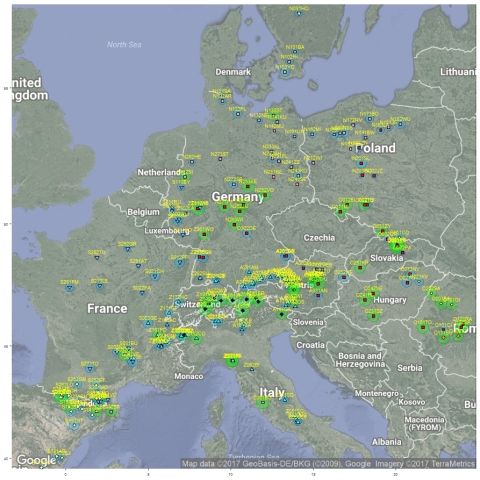
Einziger Minuspunkt (der mich persönlich aber momentan keineswegs stört): die Maschinen laufen auch bei mir nicht ununterbrochen, d.h. sie stehen manchmal still und sind ungenutzt ...
Ein Vorteil meiner eigenen (dedizierten) VMs ist die individuelle Installations- und Update-Möglichkeit von Programmen, sowohl unter Windows als auch unter Linux. Somit besteht kein Nachteil mehr gegenüber Standalone - Maschinen.“
„Auswertung im Anschluß an die NGS-AmpliconDuo-Pipeline von Angela Lange für Amplicons: bei mir u.a. NGS-Sequenzen-Clustering mit Software SWARM, parallelisiert unter Linux, mit teilweise großen csv Tabellen, d.h. > 1 Millionen Sequenzen, anschließend taxonomische Zuordnung entsprechend den Sequenz-Datenbanken NCBI (für Eukaryoten) oder PR2(für Eukaryoten) und SILVA (für Bakterien) mit Hilfe von BLASTN (basic local alignment search tool). Unter Windows: Verwenden einer 50-60 GB großen RAMDISK, dadurch wird das Allinieren zu lokal gespeicherten NCBI-Datenbank Sequenzen deutlich beschleunigt (Faktor 10-12), d.h. bei 100000 Sequenzen kann das Nadelöhr BLASTN oft in nur 1 Nacht durchlaufen, bei voller Auslastung (>98%) von 16 cores.
Filtern und statistische Auswertungen der erhaltenen NGS-Amplicon Tabellen mit Hilfe von R-Scripten, d.h. Einsatz von R, RStudio mit diversen R-packages.
Statistische Auswertungen von Transscriptom Tabellen mit Hilfe von R-Scripten.“
Anwendungen: R
Linux/Windows-VM (Ubuntu 64Bit/Eng): 16 Cores