Forschung

Strukturaufklärung von Proteinen
Das Institut für Strukturelle und Medizinische Biochemie beschäftigt sich mit der Wechselwirkung von Proteinen untereinander und der Bildung von Ligand-Protein-Komplexen. Hierzu verwenden wir verschiedene biochemische und biophysikalische Techniken. Dabei steht die Aufklärung der dreidimensionalen Strukturen von vorwiegend humanen Proteinen und deren Komplexen mit Hilfe der Kernspin-Resonanz-Spektroskopie (NMR) im Vordergrund. Unser primäres Interesse gilt dabei Enzymen, die an der post-translationalen Veränderung von Proteinen beteiligt sind.

Biochemische Charakterisierung von Parvulinen
In unseren biochemischen und molekularbiologischen Arbeiten beschäftigen wir uns mit Enzymen der Parvulinfamilie, einer Gruppe von Peptidyl-Prolyl-cis/trans Isomerasen. Das menschliche Genom kodiert für zwei dieser Proteine: Pin1 beeinflusst die Aktivität und Lokalisation von Phosphoproteinen in Zellen, während das verwandte Par14 und seine Isoform Par17 in Vorgängen wie dem Umbau des Chromatins, der RNA-Prozessierung und in Rezeptorabhängigen zellulären Prozessen (Par14) sowie bei der Bildung des Zytoskeletts und an mitochondrialen Prozessen (Par17) beteiligt sind. Zudem interessieren wir uns für die Funktion und Biochemie von Parvulinen aus einzelligen parasitären Organismen wie den Trypanosomen.

Biophysikalische Untersuchungen zur Wechselwirkung synthetischer, supramolekularer Liganden mit Proteinen
In Zusammenarbeit mit chemisch orientierten Arbeitsgruppen untersuchen wir die Wechselwirkung von Proteinen mit supramolekularen Liganden wie „organischen Pinzetten“ und kurzen, verzweigten Polymeren. Wir wollen dabei die physikochemischen Bedingungen herausfinden, unter denen eine gezielte Bindung stattfinden kann. Dies soll helfen, synthetisch optimierte Liganden herzustellen, die in der Lage sind, Protein-Protein-Wechselwirkungen gezielt zu (de-)stabilisieren oder in die Komplexbildung modulierend einzugreifen.
- Sonderforschungsbereich 1093 Supramolekulare Chemie an Proteinen (2014 - 2022)
- SFB/Transregio 60: Immuntherapie bei chronischen Virusinfektionen (2010 - 2013)
- GRK-1431: Transcription, Chromatin Structure and DNA Repair in Development and Differentiation (2006 - 2016)
- BMBF Projekt "Structure based design of MRI Probe Molecules for the highly sensitive detection of metastases.” (2009 - 2011)
Einträge in Datenbanken - NMR
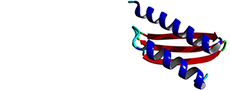
2N84
Solution structure of the FHA domain of TbPar42
Rehic E, Hoenig D, Kamba BE, Goehring A, Hofmann E, Gasper R, Matena A, Bayer P.(2019) Biomolecules 7;9(3).

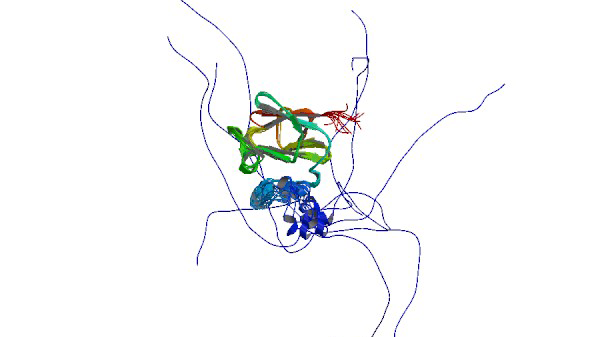
2N87
Solution structure of the PPIase domain of TbPar42
Rehic E, Hoenig D, Kamba BE, Goehring A, Hofmann E, Gasper R, Matena A, Bayer P.(2019) Biomolecules 7;9(3).
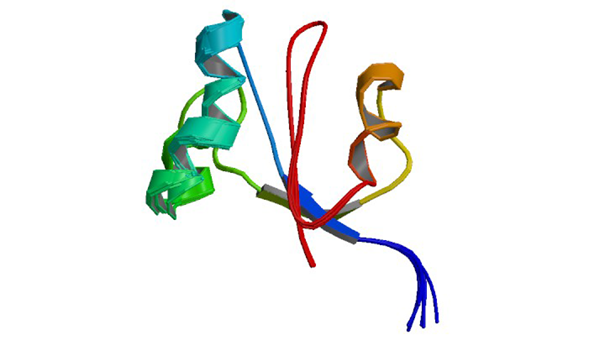
2M08
The solution structure of NmPin, the parvuline of Nitrosopumilus maritimus
Hoppstock L, Trusch F, Lederer C, van West P, Koenneke M, Bayer P. (2016) Bmc Biol. 14: 53-53.
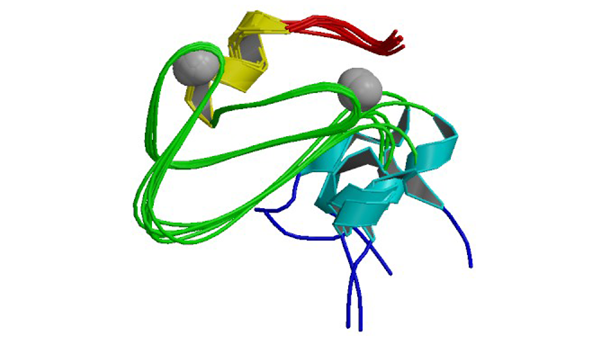
2FNF
C1 domain of Nore1
Harjes E, Harjes S, Wohlgemuth S, Müller KH, Krieger E, Herrmann C, Bayer P. (2006) Structure 14: 881-888.
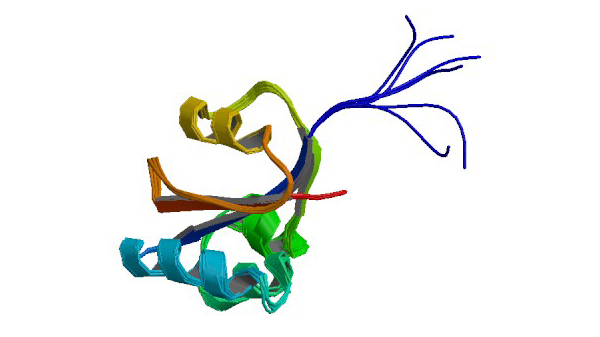
2RQS
3D structure of Pin from the psychrophilic archeon Cenarcheaum symbiosum (CsPin)
Jaremko Ł, Jaremko M, Elfaki I, Mueller JW, Ejchart A, Bayer P, Zhukov I. J. Biol.Chem. 286: 6554-6565.

2K76
Solution structure of a paralog-specific Mena binder by NMR
Link NM, Hunke C, Mueller JW, Eichler J, Bayer P. (2009) Biol.Chem. 390: 417-426.
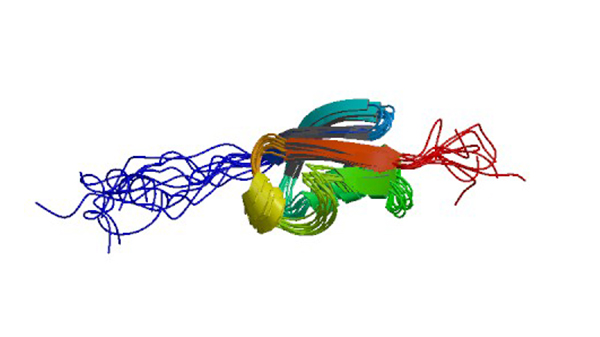
1A5R
Structure determination of the small Ubiquitin-related modifier Sumo-1, NMR, 10 Structures
Bayer P, Arndt A, Metzger S, Mahajan R, Melchior F, Jaenicke R, Becker J. (1998) J. Mol.Biol. 280: 275-286.
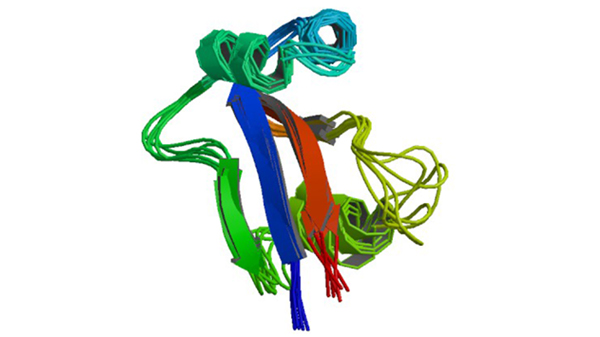
1EQ3
NMR Structure of Human Parvulin HPAR14
Sekerina E, Rahfeld JU, Müller J, Fanghänel J, Rascher C, Fischer G, Bayer P. (2000) J.Mol.Biol. 301: 1003-1017.
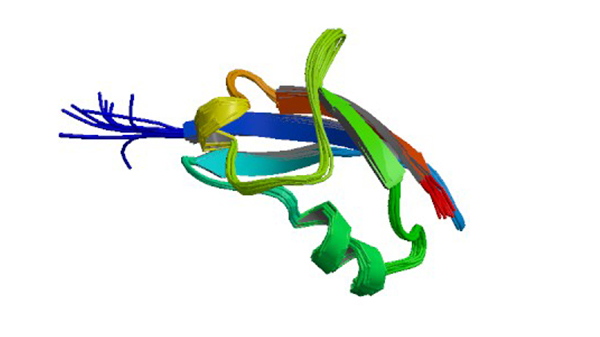
1RLF
Structure determination of the RAS-binding domain of the RAL-specific guanine nucleotide exchange factor RLF
Esser D, Bauer B, Wolthuis RM, Wittinghofer A, Cool RH, Bayer P. (1998) Biochemistry 37: 13453-13462.
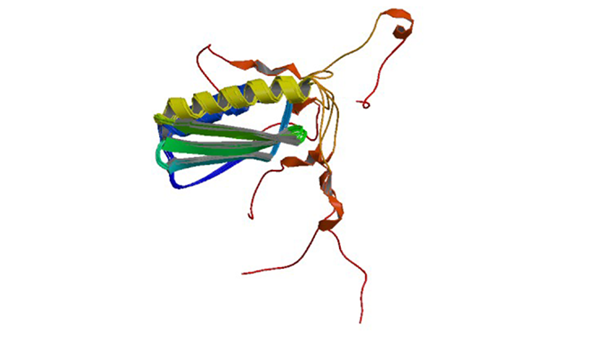
1N3G
Solution structure of the ribosome-associated cold shock response protein Yfia of Escherichia coli
Rak A, Kalinin A, Shcherbakov D, Bayer P. (2002) Biochem. Biophys.Res.Commun. 299: 710-714.
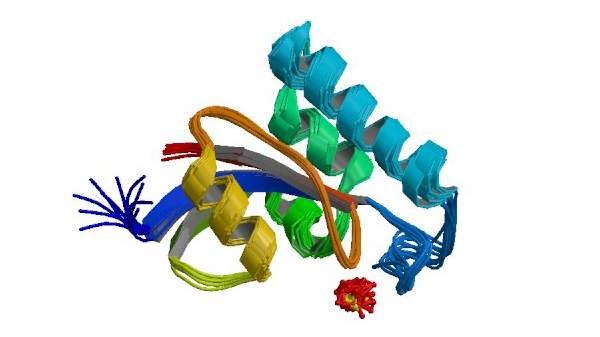
1NMW
Solution structure of the PPIase domain of human Pin1
Bayer E, Goettsch S, Mueller JW, Griewel B, Guiberman E, Mayr LM, Bayer P. (2003) J. Biol. Chem. 278: 26183-26193.
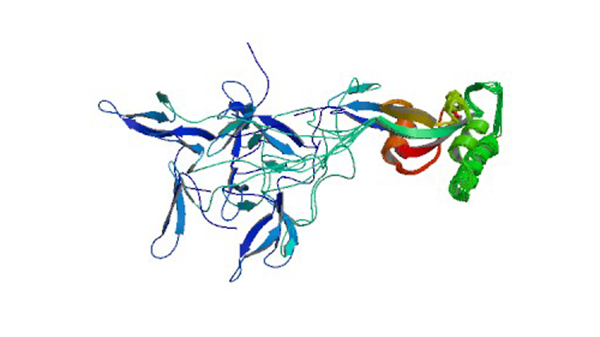
1NMV
Solution structure of human Pin1
Bayer E, Goettsch S, Mueller JW, Griewel B, Guiberman E, Mayr LM, Bayer P. (2003) J. Biol. Chem. 278: 26183-26193.
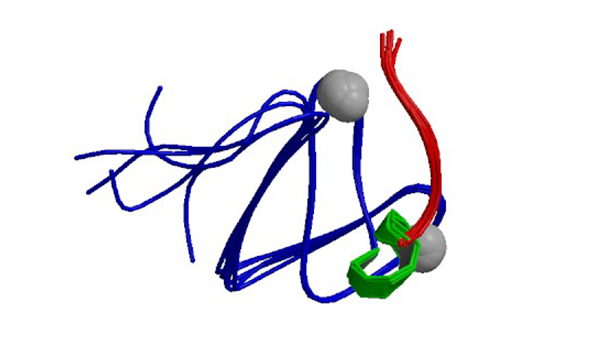
1RFH
Solution structure of the C1 domain of Nore1, a novel Ras effector
Harjes E, Harjes S, Wohlgemuth S, Müller KH, Krieger E, Herrmann C, Bayer P. (2006) Structure. 14(5):881-8.
Einträge in Datenbanken - X-Ray
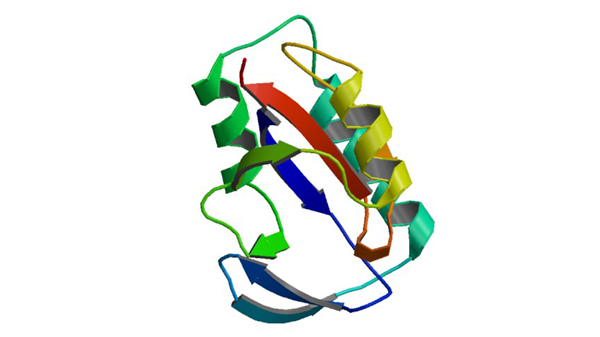
6GMP
Crystal Structure of the PPIASE Domain of TBPAR42
Rehic E, Hoenig D, Kamba BE, Goehring A, Hofmann E, Gasper R, Matena A, Bayer P. Biomolecules. 2019 7;9(3).
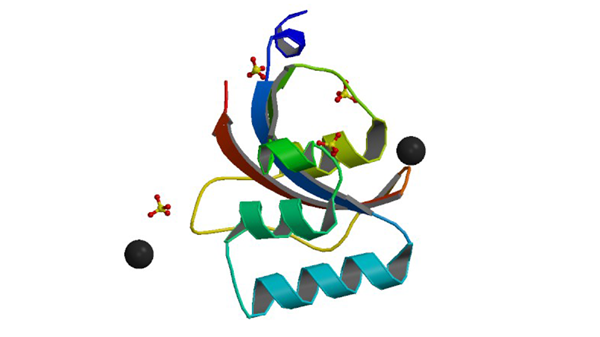
3UI4
0.8 A resolution crystal structure of human Parvulin 14
Mueller JW, Link NM, Matena A, Hoppstock L, Rüppel A, Bayer P, Blankenfeldt W. (2011) J. Am. Chem. Soc. 133: 20096-20099.
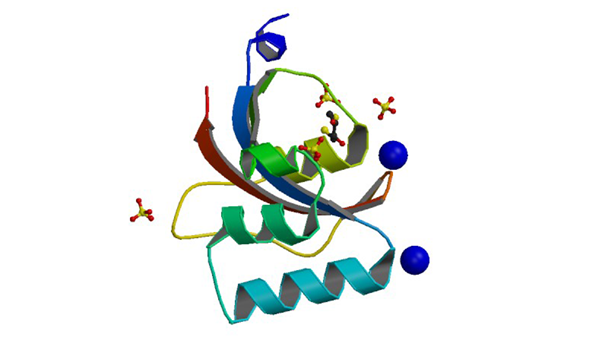
3UI5
Crystal structure of human Parvulin 14
Mueller JW, Link NM, Matena A, Hoppstock L, Rüppel A, Bayer P, Blankenfeldt W. J. Am. Chem. Soc. 133: 20096-20099.
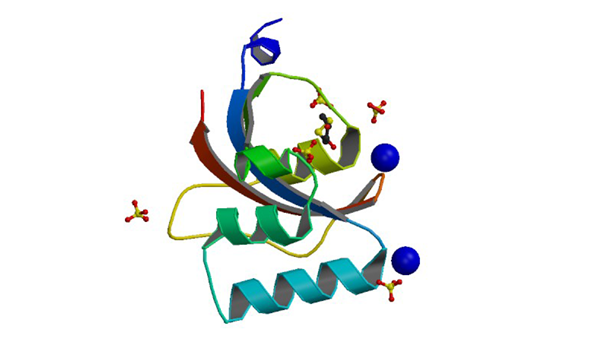
3UI6
0.89 A resolution crystal structure of human Parvulin 14 in complex with oxidized DTT
Mueller JW, Link NM, Matena A, Hoppstock L, Rüppel A, Bayer P, Blankenfeldt W. J. Am. Chem. Soc. 133: 20096-20099.
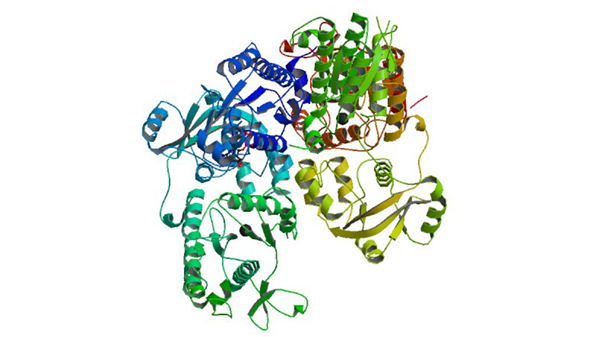
1X6V
The crystal structure of human 3'-phosphoadenosine-5'-phosphosulfate synthetase 1
Harjes S, Bayer P, Scheidig AJ. (2005) J. Mol. Biol. 347: 623-635.
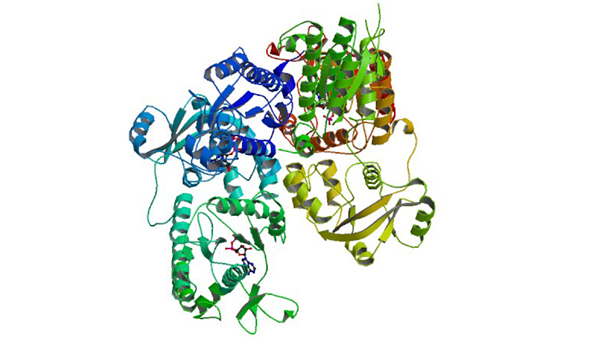
1XJQ
ADP Complex OF HUMAN PAPS SYNTHETASE 1
Harjes S, Bayer P, Scheidig AJ. (2005) J. Mol. Biol. 347: 623-635.
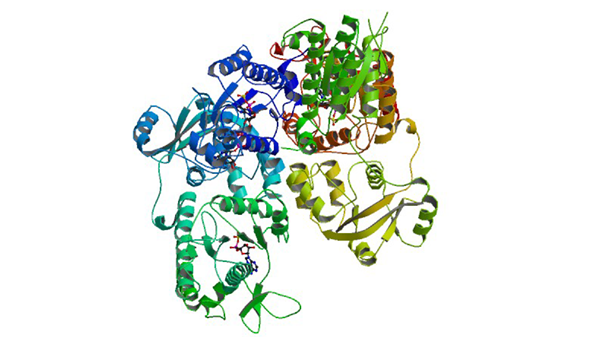
1XNJ
APS complex of human PAPS synthetase 1
Harjes S, Bayer P, Scheidig AJ. (2005) J. Mol. Biol. 347: 623-635.
BMRB Datenbank:
BMRB-11080
BMRB-15946
BMRB-ID 4768
BMRB-ID 4768
BMRB-ID 5979
BMRB-ID 5979
BMRB-ID 6059
BMRB-ID 6059
Das regionale Netzwerk bio-N3MR ist ein loser Zusammenschluss von Forschergruppen aus Nordrhein-Westfalen, welche mit Hilfe der kernmagnetischen Resonanzspektroskopie (NMR) biologisch relevante Makromoleküle untersuchen. Ziele unserer Arbeit sind ein tieferes Verständnis biologischer Phänomene auf atomarer Ebene, das Verstehen des Zusammenspiels von Biomolekülen bei komplexen biologischen Prozessen, die Aufklärung der molekularen Ursachen und Mechanismen krankhafter Veränderungen in Organismen und die Suche nach neuartigen Wirkprinzipien und Wirkstoffen für die Bekämpfung von Krankheiten.